- A+
今天分享一篇发表在ACS Sensors(2021年IF=7.711)上的文章,名为《DNA Nanowire Guided-Catalyzed Hairpin Assembly Nanoprobe for In Situ Profifiling of Circulating Extracellular Vesicle-Associated MicroRNAs》[1](DNA纳米线引导催化发夹组装纳米探针用于循环胞外囊泡相关microRNA)。
细胞外囊泡(EVs)是一种双层脂质包裹的膜囊泡,直径从50到200nm,几乎所有类型的细胞都会分泌。它们可以通过囊泡内生物分子的转移,将生物信息传递到目标细胞。MicroRNAs(miRNAs)是一组可以大量封装在EV(EV-miRNAs)中的小RNA。它们作为基因调节因子参与多种生命过程,并与许多疾病相关。由于脂质膜的保护作用,EV-miRNAs可以在血液和其他体液中稳定检测到,这使它们有望成为一种新型的生物标志物。
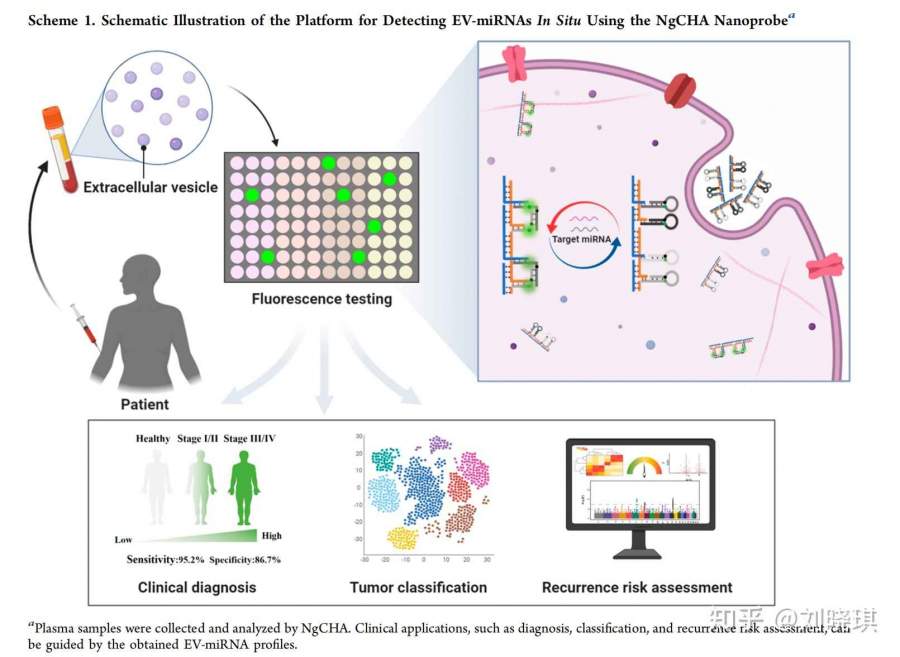
目前的EV-miRNAs分析通常需要EV裂解和总RNA提取,然后进行逆转录和定量聚合酶链反应(qPCR)分析。然而,这些方法还远不能令人满意。除了长时间的处理时间和复杂的操作,实验所需的EV裂解导致不同程度的RNA降解。此外,多个实验步骤会导致RNA的丢失,检测结果的重现性较差,这阻碍了EV-miRNAs的准确定量。EV-miRNAs的原位分析可能为解决上述缺点提供了另一种策略。通过将特定的探针加载到单个EV中,可以直接从EV内的靶向miRNA中检测到信号,从而避免EV裂解和RNA提取。
有效将检测探针运送到纳米级EV中,和与目标结合后的信号放大是该策略的两个主要挑战。目前使用脂质体进行膜融合或通过物理或化学方法进行膜穿孔的传递方法已经被提出。然而,脂质体是不稳定的,嵌入的货物在进入EV之前就被释放了。基于穿孔的方法可能会损伤EV膜,并在检测前导致EV-miRNA泄漏。在信号输出方面,常用探针如分子信标(MBs)或纳米片的放大效率较低,导致EV内的信号相对较弱,限制了其分析灵敏度。
DNA纳米线是由互补序列组成的纳米级结构。作为一类一维生物材料,它们可以很容易地被编程并连接到核酸扩增系统上。此外,由于DNA的粘性末端被包裹在DNA纳米线内,它们可以在生物环境中避免被核酸酶降解。在这里,我们提出了一种DNA纳米线引导催化的发夹组装(NgCHA)系统,用于原位EV-miRNA的检测。该策略结合了DNA纳米线的高穿透率和改进的催化发夹组装(CHA)扩增系统在紧凑空间内的信号增强,允许快速感知EV-miRNA原位,其性能优于传统的CHA和MB探针。使用EV-miRNA面板(miR-375、miR-1246、miR-221和miR-21),该系统可以区分癌症和非癌症患者,并通过血液样本帮助进行疾病监测(方案1),这显示了其作为EV分析的非侵入性工具的潜力。
研究结果:
一、NgCHA纳米探针的组装与表征
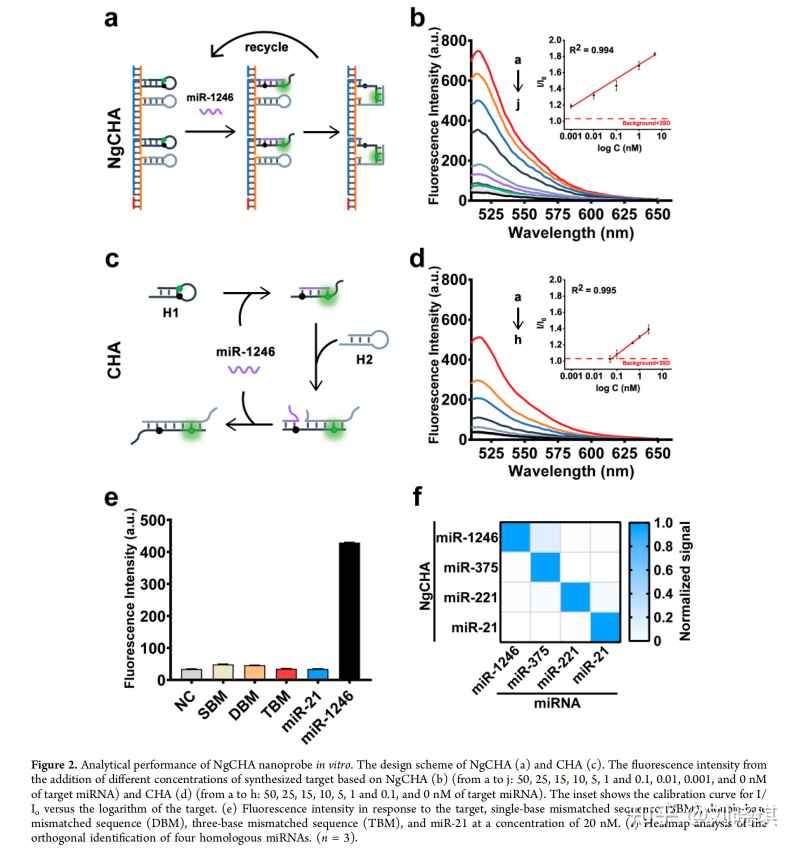
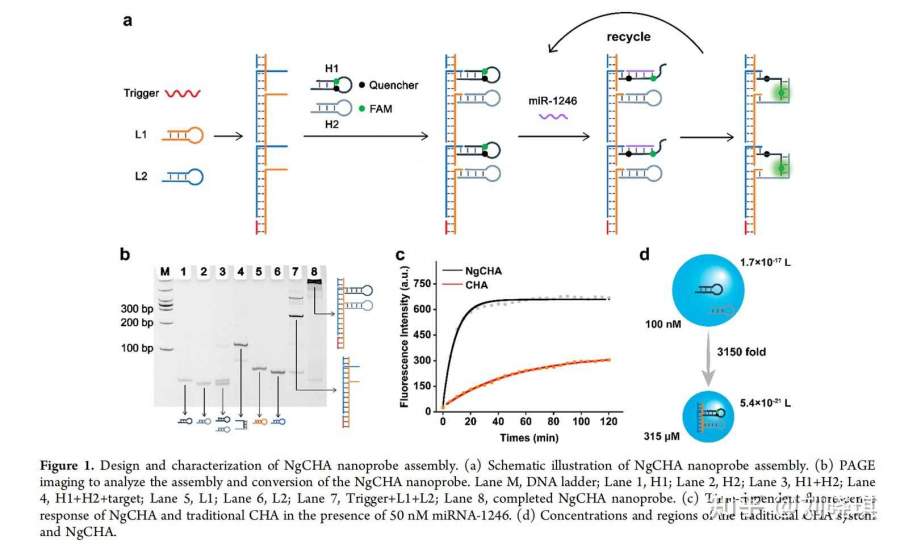
为了证明该平台的原理,我们选择了miRNA-1246作为模型,该模型在癌症患者的血浆EV中表达上调。NgCHA纳米探针与DNA纳米线和CHA系统集成(图1a)。在一个典型的CHA反应中,靶miRNA触发了两个发夹(H1和H2)的趾点链置换组装,从而导致靶miRNA和CHA产物的循环重用(图2c)。在这项工作中,纳米线由触发器L1和L2以摩尔比为1:2:2自组装,这确保了最小长度的更好的EV穿透。CHA的两个发夹被固定在纳米线上。然后,将CHA体系(H1和H2)在纳米线上交替排列,形成NgCHA纳米探针。H1的茎被荧光素酰胺(FAM)和猝灭剂(BHQ1)标记,当发夹打开时,可以产生荧光信号。在加入靶miRNA后,CHA可以沿着纳米线启动。
采用聚丙烯酰胺凝胶电泳(PAGE)对NgCHA的组装过程进行了表征。如图1b所示,CHA可以通过合成的靶标miRNA(Lane4)和少量的背景信号(Lane3)成功启动。将单序列(Lane 1,H1; Lane 2,H2; Lane 5,L1; Lane 6,L2)组合成DNA纳米线(Lane 7)和完整的NgCHA(Lane 8)。在Lane 7中,大部分形成了长度约为250bp的DNA纳米线,并产生了一些长度在250bp以上的较长的DNA纳米线。用琼脂糖凝胶电泳检测NgCHA的纯度。如图S1所示,大部分NgCHA纳米探针显示在长度约为380bp的条带上,在380bp以上形成了一些较长的NgCHA分子。这些结果证明了NgCHA探针的成功组装和可接受的纯度,并证实了其检测miRNAs的可行性。
采用随时间变化的荧光信号试验,研究了均相溶液中的反应动力学。加入25nM合成的miRNA-1246后,在515nm处检测了120min后FAM的荧光恢复率(图 1c)。NgCHA产生的时间依赖性信号增加,直到在30min(黑线)达到平台期,远短于CHA(90min)。此外,NgCHA显示出高2.2倍的荧光信号。这些结果表明,NgCHA纳米探针具有较高的灵敏度和效率。
NgCHA较高的荧光信号可以用碰撞理论来解释:根据公式V=1/cN(c代表发夹的浓度,N是阿伏加德罗常数),当典型的CHA发夹浓度为100nM时,可反应的两个发夹之间最近距离形成的球体体积为1.7×10^(?17)L。在NgCHA系统中,DNA纳米线中H1和H2发夹之间的距离减少到32bp(10.88nm)。在DNA纳米线的约束下,最小反应体积为5.4×10^(?21)L,CHA的浓度提高到315μM。其反应效率是传统CHA的3150倍(图 1d)。因此,被DNA纳米线局部固定的h1和h2比CHA的碰撞更频繁,提高了反应效率和灵敏度。
二、NgCHA对合成目标的分析性能
H1和H2之间的距离在CHA呼吸反应中起着至关重要的作用,可能是背景信号的主要原因。因此,我们测试了H1和H2之间的不同距离,并选择了32bp的最佳距离。然后研究了NgCHA和CHA的检测性能。NgCHA的荧光强度逐渐增加,增加目标从1p M到50nm的检测极限(LOD)0.8p M(图2c,d),比CHA低108倍(图2e,f),比分子信标低8493倍。为了评估NgCHA的特异性,我们检测了一个单碱基错配序列(SBM)、一个双碱基错配序列(DBM)、一个三碱基错配序列(TBM)和一个完整的错配序列(miRNA-21)。如图2g所示,只有miRNA-1246可以启动该反应。对于癌症分析,通常需要多个miRNA;因此,我们进一步评估了NgCHA在多重miRNA中的分析性能。我们设计了4个NgCHA纳米探针用于miRNAs的正交鉴定(miR-221、miR-375、miR-1246和miR-21)(图2h)。NgCHA在添加靶miRNAs时产生的信号要比在添加非靶miRNAs时产生的信号要高得多。这些结果证明了NgCHA的多功能性,并支持其用于进一步的癌症管理。接下来,通过在实验室内和批间试验中检测1nM和0.01nM的miR-1246样品,来评价该方法的重现性。批内和批间变量系数分别为1.83%和5.89%。这些结果表明,该荧光平台具有出色的重现性。通过与阴性的人血浆进行孵育,验证了该探针的稳定性。荧光试验和琼脂糖电泳试验结果均表明,NgCHA纳米探针比传统的DNA探针更稳定,这可能是由于DNA纳米线对DNA探针的保护作用。
三、NgCHA原位检测EVmiRNA的分析性能
采用标准超离心法从三种不同的乳腺癌(BC)细胞系(MDA-MB-231、MCF-7、SK-BR3)和一个良性乳腺上皮细胞系(MCF-10A)中提取的EV用于检测。透射电子显微镜(TEM)图像展示了一个具有凹形的椭圆形EV结构。Western blot分析显示了四种EV蛋白(CD9、CD81、CD63和TSG101)的特异性表达。纳米颗粒跟踪分析(NTA)显示,颗粒粒径在76~225nm之间,置信度为95%。这些结果与文献中报道的EV的结果一致,表明所获得的EV是完整的,可用于接下来的实验。
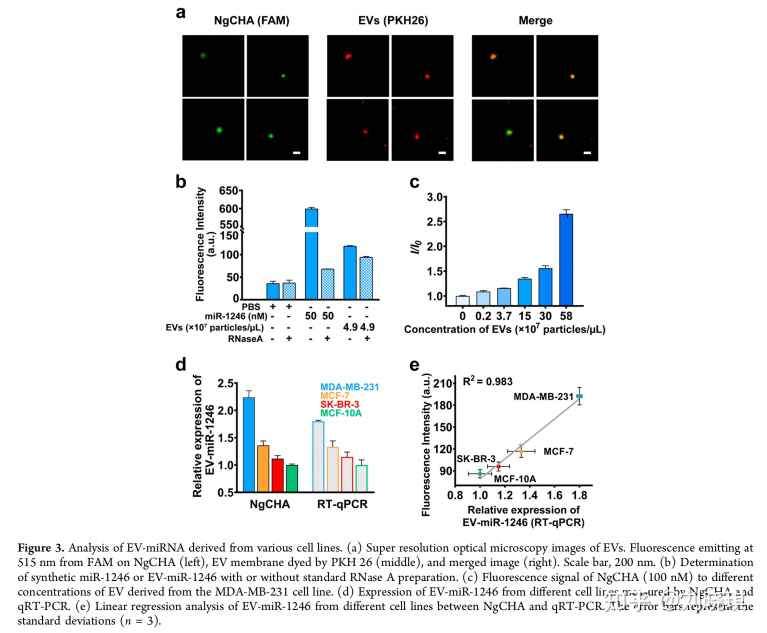
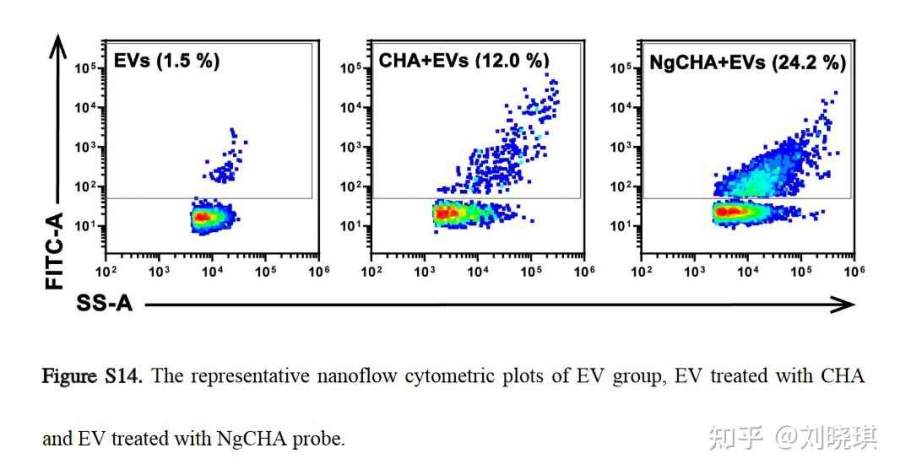
为了证实NgCHA进行原位分析的可行性,我们将NgCHA与不同浓度的MDAMB-231细胞来源的EV一起孵育。加入EV后,荧光信号逐渐增加,这显示了随机纳米探针的差异。利用超分辨率荧光显微镜对NgCHA(FAM)和EV(PKH26)的共定位也表明,NgCHA纳米探针进入了EV管腔(图3a)。为了进一步确认荧光信号的来源,我们采用了国际细胞外囊泡学会(ISEV)推荐的样品制备方法。用蛋白酶K和RNase完全降解miRNA和蛋白?miRNA复合物,它们可能结合EV表面。合成miR-1246的NgCHA探针产生的荧光信号在样品处理后下降到阴性对照水平。当使用MDA-MB-231细胞来源的EV的样品制备时,与未处理的EV相比,荧光强度略有下降(图3b)。这些结果进一步证明了NgCHA纳米探针可用于直接检测EV-miRNA。此外,荧光信号与EV浓度明显相关,在浓度低至2×106颗粒/μL时,与空白探针有显著差异(图3c)。NgCHA孵育的EV的代表性的纳米流式(NanoFCM)分析显示,信号明显强于其他组(EV组,EV与CHA孵育的组)。这些结果证明了NgCHA纳米探针对原位检测EV-miRNA的灵敏度。以下原因可能与NgCHA的内化机制有关。首先,NgCHA的刚性结构可以大大提高细胞的摄取效率,这已经在之前的报道中得到了证实。其次,NgCHA纳米探针的角落可以基于“类电荷吸引”机制促进其传递。
为了研究该平台的检测准确性,我们用NgCHA纳米探针检测了来自MDAMB-231、MCF-7、SK-BR3和MCF-10A细胞系的EV中EV-miRNAs的水平和实时(RT)-qPCR。来自MDA-MB-231细胞系的EV的荧光信号比来自MCF-7、SK-BR-3和MCF-10A细胞的EV的荧光信号分别高约1.6倍、2.0倍和2.2倍(图3d)。此外,线性回归分析表明,NgCHA结果与RT-qPCR获得的数据高度成正比,相关系数为0.983(图3e)。
四、四种用于BC诊断和复发风险评估的EV-miRNA图谱
接下来,我们用真实的临床样本评估了该平台的性能。我们选择了一些假定的与肿瘤相关的EV-miRNA来在EV上进行检测。MiR-21是一种癌症生物标志物,因为它在各种癌症中都表达上调。研究表明,miRNA-21通过抑制肿瘤抑制基因来促进肿瘤进展。miR-1246也被发现在乳腺癌、结肠直肠癌、胰腺癌、卵巢癌和其他癌症中过表达。它在促进肿瘤转移中起着关键作用。MiR-221参与抑制基因表达,如细胞周期蛋白依赖性激酶抑制剂p27和雌激素受体(ER),通过干扰正常的细胞增殖过程导致肿瘤的发展。MiR-375也被确定为ER的调节因子和治疗的潜在预测因子。miR-375的高表达可通过抑制p27和ER而导致肿瘤的发展。在公共数据库验证后,我们使用了4-EV-miRNAs(miR-221、miR-375、miR-1246和miR-21)作为我们的候选生物标志物,因为它们在各种肿瘤中的高表达。
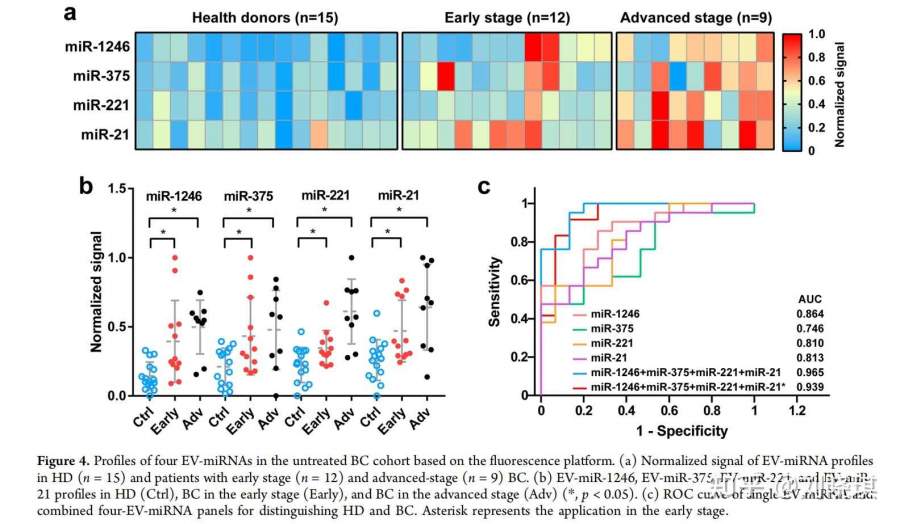
然后,我们测试了所提出的平台是否可以用于BC诊断。共收集了36例未经治疗的BC患者(n=21,早期=12,晚期=9)和健康供体(HD)(n=15)的血浆样本,并通过4个EV-miRNA面板进行分析。如图4a所示,四种EV-miRNA在不同患者中的表达水平差异很大。根据从36个临床样本中测量的4个EV-miRNAs的散射图(图4b),对于BC患者和HD,EV-miR-1246、EV-miR-375、EV-miR-221和EV-miR-21的差异明显。证明了EV-miRNA可用于区分BC和HD。采用受试者工作特征(ROC)曲线分析来评估个体EV-miRNAs区分BC患者和HDs的能力。如图4c所示,EV-miRNAs组合后的曲线下面积(AUC)显著改善,而EV-miR-1246、EV-miR-375、EV-miR-221和EV-miR-21的AUC分别为0.894、0.746、0.810和0.875。与单一EV-miRNA和其他EV-miRNA组合相比,4种EV-miRNA组合提供了最好的准确性(AUC:0.965,95%CI:0.942-1.000)(图4c)。值得注意的是,4种EV-miRNAs联合治疗在乳腺癌早期的AUC为0.945,敏感性和特异性分别为95%和100%(图4c),提示所提出的BC诊断平台具有较高的临床可行性。
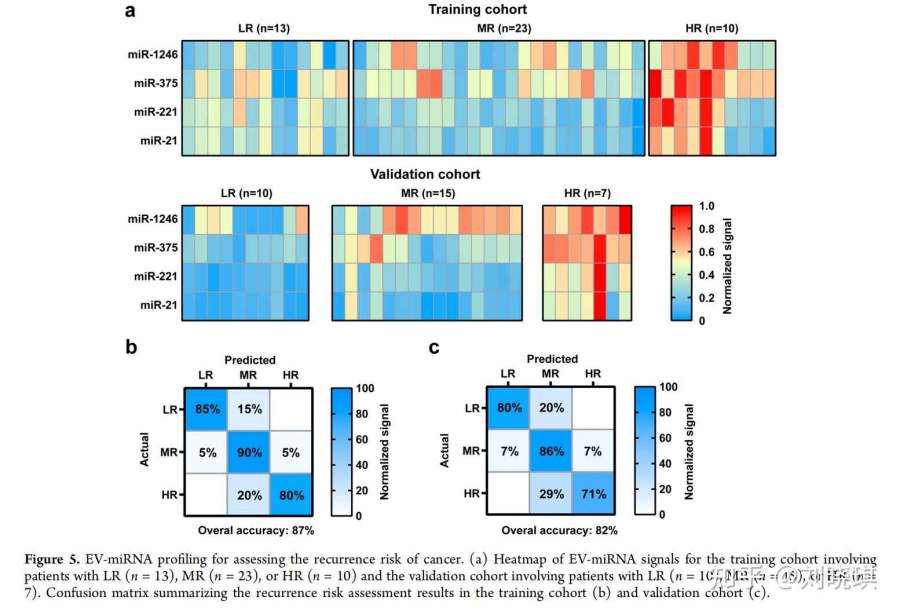
手术治疗是BC患者的主要治疗方法之一。手术治疗后,通过临床综合分析来评估BC患者的复发风险,确定对患者的随访治疗方案。因此,我们评估了EV-miRNA分析结合随机森林算法(RF)的能力,这是一种新兴的和高度灵活的机器学习算法,以评估从手术治疗后收集的BC患者的78个血浆样本的复发风险。图5a总结了高危(HR)(n=23)、中风险(Mr)(n=38)和低风险(LR)(n=17)患者EV-miRNA表达水平的相对变化,这些患者均被临床估计证实。对训练队列和验证队列的复发风险评估结果进行了定量总结,形成了混淆矩阵(图5b,c)。采用RF方法进行复发风险评估,训练队列的总体准确率为87%,验证队列中为82%。EV-miR-1246在癌症分类中所占比例高于EV-miR-1246,说明EV-miRNA-1246在复发风险评估中的重要作用。
五、提出了区分多种癌症的平台
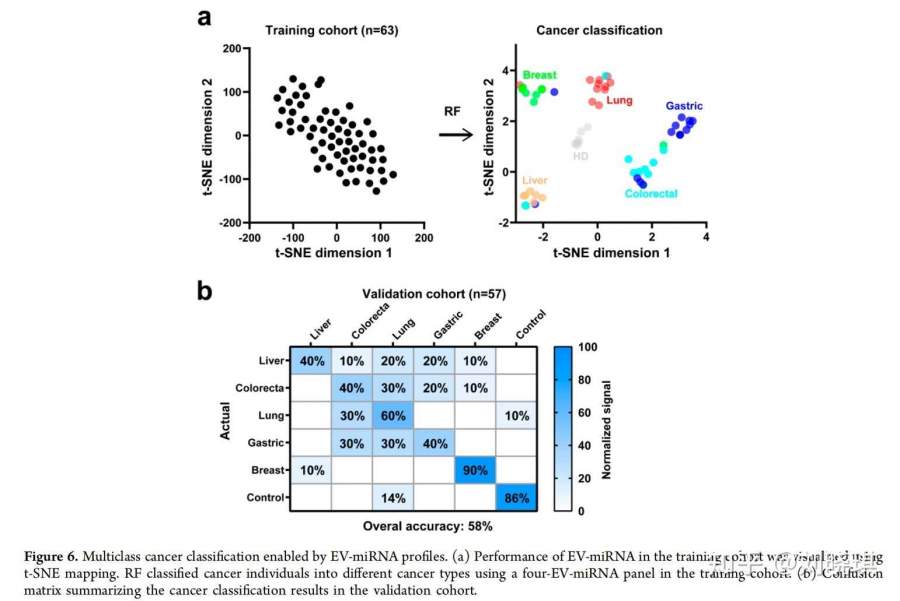
该平台在BC管理中的良好性能促使我们进一步探索其在癌症分析中的实用性。不同肿瘤来源的治疗方法不同,目前的方法很难区分晚期患者的肿瘤来源。因此,识别肿瘤类型对于评价肿瘤来源和有效治疗肿瘤具有重要价值。在这项工作中,我们使用EV-miRNA面板结合射频验证肿瘤类型分化在临床队列覆盖一系列肿瘤类型的可能性,包括BC(n=21例),肝癌(n=21例),肺癌(n=21例),胃癌(n=21例)和结直肠癌(n=21例)。EV-miRNA图谱在训练队列中通过RF进行处理,并在验证队列中得到证实。通过使用t-SNE将RF的性能映射到一个二维平面上,我们发现大多数不同类型的肿瘤都是分离的(图6a)。根据基于训练队列和验证队列(图6b)的混淆矩阵,该模型在识别BC和肺癌方面具有较好的准确性,而对肝癌和结肠直肠癌的识别准确性较低。在多类癌症诊断中,训练队列的总体准确率为62%,验证队列的总体准确率为58%,优于验证队列中的其他机器学习算法(NN35%,SVM32%,LDA38%)。它的准确性也与之前报道的8-EV蛋白面板相当。这些结果表明,所提出的模型能够很好地区分肿瘤类型。虽然EV-miR-1246具有最高的相对重要性,但其他EV-miRNA的相对重要性不低于20%,表明了每个EV-miRNA在多类癌症诊断中不可或缺的作用。因此,EV-miRNA谱结合RF可以帮助分类癌症类型,支持疾病诊断,从而对原发肿瘤来源不明的癌症患者实现更有效的治疗。
总结:
总的来说,我们开发了一种新型的NgCHA纳米探针,用于原位分析EV-miRNA,并展示了其在临床样本中的应用。通过将DNA纳米线与CHA系统连接起来,NgCHA能够直接检测具有高稳定性和高渗透性的EV-miRNA。DNA纳米线在紧凑空间内的CHA系统通过增加局部浓度进一步提高了分子碰撞概率,具有比传统CHA和MB纳米探针的灵敏度。通过EV-miRNA面板的结合,我们发现可以成功地实现BC诊断、复发风险评估和多种癌症的鉴别。与目前的RT-qPCR方法需要操作复杂、终点检测相比,该方法具有操作简单、实时监测、成本低等优点。此外,它还可与临床实验室广泛使用的常用酶标仪集成。它也可以很容易地用不同的发夹进行编程,用于检测其他与疾病相关的核酸。我们假设这种方法在基于EV的疾病管理中具有很大的潜力。
参考文献:
[1]Zhang Ye,Wu Yuan,Luo Shihua,et al.DNA Nanowire Guided-Catalyzed Hairpin Assembly Nanoprobe for In Situ Profiling of Circulating Extracellular Vesicle-Associated MicroRNAs.ACS sensors.2022;7 (4):1075-1085.doi:10.1021/acssensors.1c02717