- A+
随着各国大规模基因组计划的推进(如“十万人基因组计划”、“万鸟计划(B10K)”、“Zoonomia联盟”、“拟南芥1001基因组计划” … …),比较基因组学终于迎来了她的黄金时代。而最近Nature上3篇比较基因组学文章的同时发表(详见文末参考文献),再一次把比较基因组学带到了它的高光时刻,比较基因组的黄金时代真的已经来了!!!
然而,对于刚接触基因组学的老师和同学来说,在做比较基因组时经常会遇到一些困惑:物种这么多,我应该选择哪些物种?选择多少个物种比较合适?比较基因组比的是基因组信息还是蛋白信息?怎样查询我想选择的物种的相关信息呢?… …
为帮助小伙伴们扫清这些科研障碍,今天小欧为大家梳理了这篇入门教程,几个步骤助您少走冤枉路,快速锁定候选物种,并顺利获取候选物种的基因组信息。
· 首先,根据关注点,划定物种选择范围。
选择物种前,首先明确自己想通过比较基因组分析探究哪些生物学问题,关注点不同,选择物种的范围也不同。比如Zoonomia联盟发表在Nature上的文章中,研究者关注的是物种尽量覆盖全面,因此,在进行物种选择时主要遵循了两个原则:①寻求最大化进化分支的长度,真兽亚纲哺乳动物每个科中至少包括一个物种;② 优先考虑医学、生物或生物多样性保护感兴趣的物种。
通过这样的物种选择设计,使收集到的具有代表性的基因组占哺乳动物科物种的82%。在此,也建议大家在选择物种时,可以先查询一下有同源或近源物种比较基因组分析的经典文献加以借鉴。
· 其次,获得候选物种的高质量基因组信息和基因组注释信息。
一套完整的比较基因组分析包括基因家族聚类、系统进化分析、分歧时间估算、基因家族收缩和扩张分析、正选择分析、基因组共线性分析以及全基因组复制事件分析,其中既要用到基因组信息也要用到基因组的注释信息(如蛋白质的氨基酸序列)。因此,做比较基因组分析的前提条件是获得候选物种的高质量(一般为染色体级别)基因组信息和基因组注释信息。
获得物种基因组信息的方法有多种,最快速、便捷的当然是利用NCBI,可以通过以下步骤进行查询。
< 第一步:选择Taxonomy
在搜索框中直接输入物种名称(可以是常用名称,也可以是拉丁文名称),或者输入想要查询的物种所在的科或属的拉丁文。我们以茄科(solanaceae)为例进行查询,可以查询到相应链接(如下图)。
随着各国大规模基因组计划的推进(如“十万人基因组计划”、“万鸟计划(B10K)”、“Zoonomia联盟”、“拟南芥1001基因组计划” … …),比较基因组学终于迎来了她的黄金时代,而最近Nature上3篇比较基因组学文章的同时发表(详见文末参考文献),再一次把比较基因组学带到了它的高光时刻,比较基因组的黄金时代真的已经来了!!!
然而,对于刚接触基因组学的老师和同学来说,在做比较基因组时经常会遇到一些困惑:物种这么多,我应该选择哪些物种?选择多少个物种比较合适?比较基因组比的是基因组信息还是蛋白信息?怎样查询我想选择的物种的相关信息呢?… …
为帮助小伙伴们扫清这些科研障碍,今天小欧为大家梳理了这篇入门教程,几个步骤助您少走冤枉路,快速锁定候选物种,并顺利获取候选物种的基因组信息。
· 首先,根据关注点,划定物种选择范围。
选择物种前,首先明确自己想通过比较基因组分析探究哪些生物学问题,关注点不同,选择物种的范围也不同。比如Zoonomia联盟发表在Nature上的文章中,研究者关注的是物种尽量覆盖全面,因此,在进行物种选择时主要遵循了两个原则:①寻求最大化进化分支的长度,真兽亚纲哺乳动物每个科中至少包括一个物种;② 优先考虑医学、生物或生物多样性保护感兴趣的物种。
通过这样的物种选择设计,使收集到的具有代表性的基因组占哺乳动物科物种的82%。在此,也建议大家在选择物种时,可以先查询一下有同源或近源物种比较基因组分析的经典文献加以借鉴。
· 其次,获得候选物种的高质量基因组信息和基因组注释信息。
一套完整的比较基因组分析包括基因家族聚类、系统进化分析、分歧时间估算、基因家族收缩和扩张分析、正选择分析、基因组共线性分析以及全基因组复制事件分析,其中既要用到基因组信息也要用到基因组的注释信息(如蛋白质的氨基酸序列)。因此,做比较基因组分析的前提条件是获得候选物种的高质量(一般为染色体级别)基因组信息和基因组注释信息。
获得物种基因组信息的方法有多种,最快速、便捷的当然是利用NCBI,可以通过以下步骤进行查询。
< 第一步:选择Taxonomy
在搜索框中直接输入物种名称(可以是常用名称,也可以是拉丁文名称),或者输入想要查询的物种所在的科或属的拉丁文。我们以茄科(solanaceae)为例进行查询,可以查询到相应链接(如下图)。
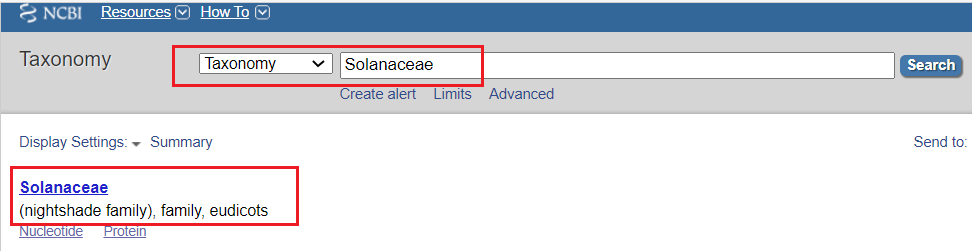
< 第二步:打开Solanaceae的链接
可以看到如下界面:
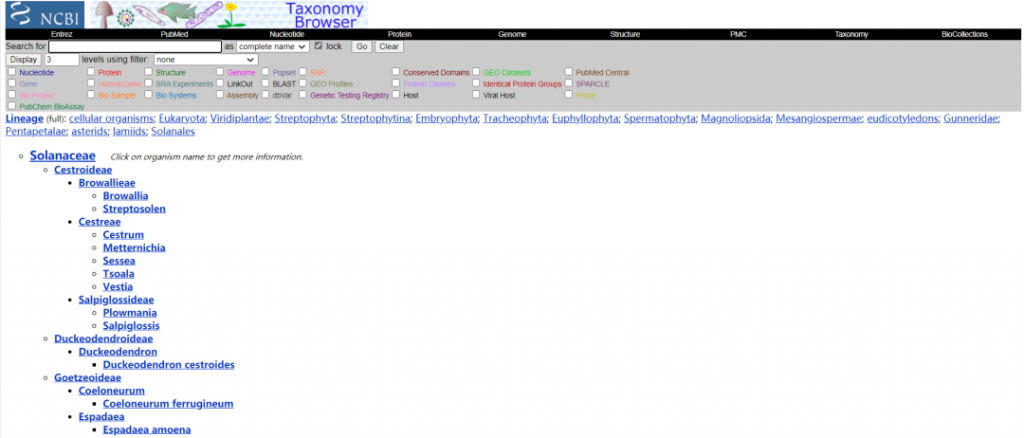
< 第三步:根据需要进行相应的筛选
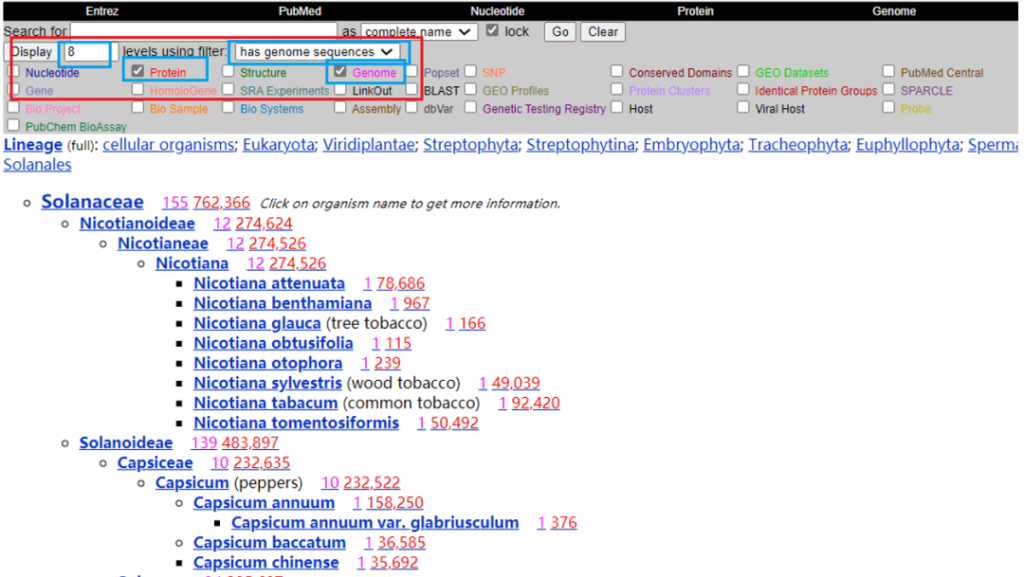
比如我们选择茄科中有基因组信息和蛋白信息的物种,如红框中的蓝框所标示,把Genome和Protein分别勾选。另外,为了全部显示有基因组的序列,将Display可展示的levels数字设置大一些(默认为3,图中设置为8),并将filter选择为“has genome sequences”(默认为none)。然后点击“Go”便可以轻松将茄科有基因组信息和蛋白信息的物种筛选出来(如下图)。最小的层级对应的是每一个物种(species),基因组信息(粉色数字)和蛋白信息(红色数字)可以直接点击链接查看。
< 第四步:查看基因组信息和基因组注释信息
我们以其中的烟草(common tobacco)为例,点进去看一下,得到下图。从这里就可以得到比较基因组分析所需要的该物种的基因组网址链接和基因组注释文件(GFF)链接(红框标示)。如果该物种已经有多个组装本,您可以通过list(蓝框标示)点击进去,选择最新的或者最经典的组装本信息。
这里要为刚入门的小伙伴们科普一下:GFF文件的全称是Generic Feature Format,是一种简单方便的对DNA、RNA以及蛋白质序列的特征进行描述的数据格式,描述的是基因组上各种特征的区间信息,由tab键隔开的9列组成,每一列代表不同的信息。GFF格式是目前序列注释的通用格式。
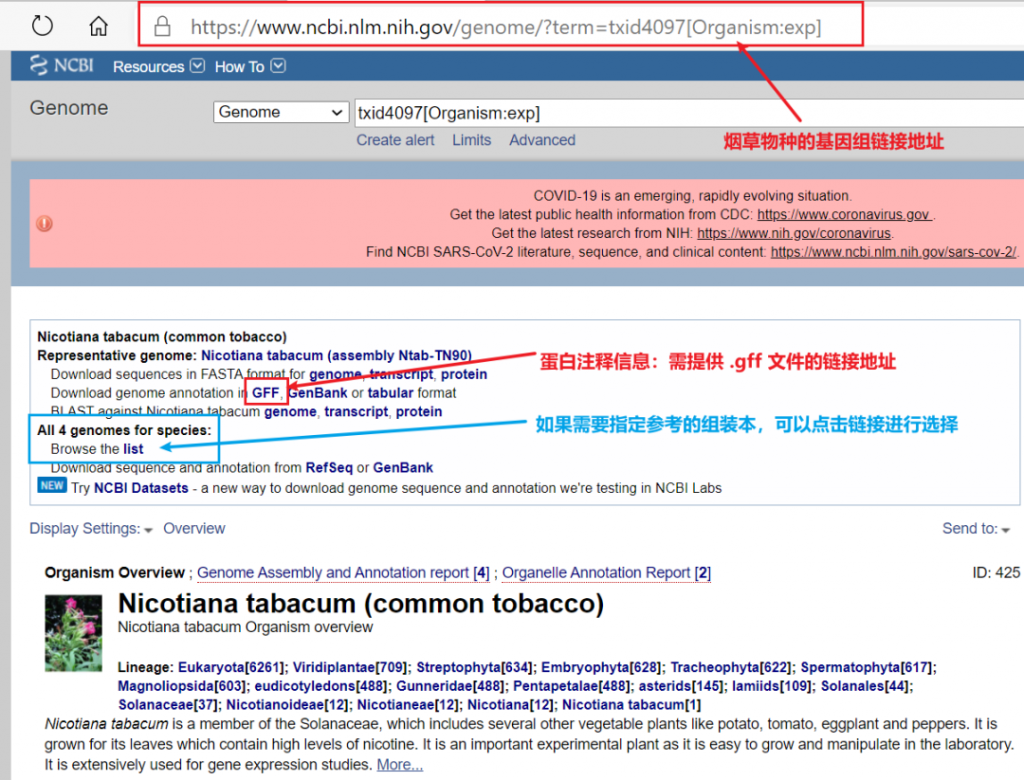
以上就是最理想的候选物种基因组和基因组注释文件的获取方式,是不是很简单?然而,当您查询时,就会明白现实与理想之间还有一步之遥——遥不可及… …

而这遥远的一步往往是因为某个非常想选择的物种的GFF注释文件找不到。比如仍然继续使用前面的例子,下图中红色箭头标示出的这几个物种,打开基因组链接,发现里面都没有GFF文件… …

(这几个物种有一个共同点:代表蛋白信息的红色数字的数值与其他物种的数值都很小。)
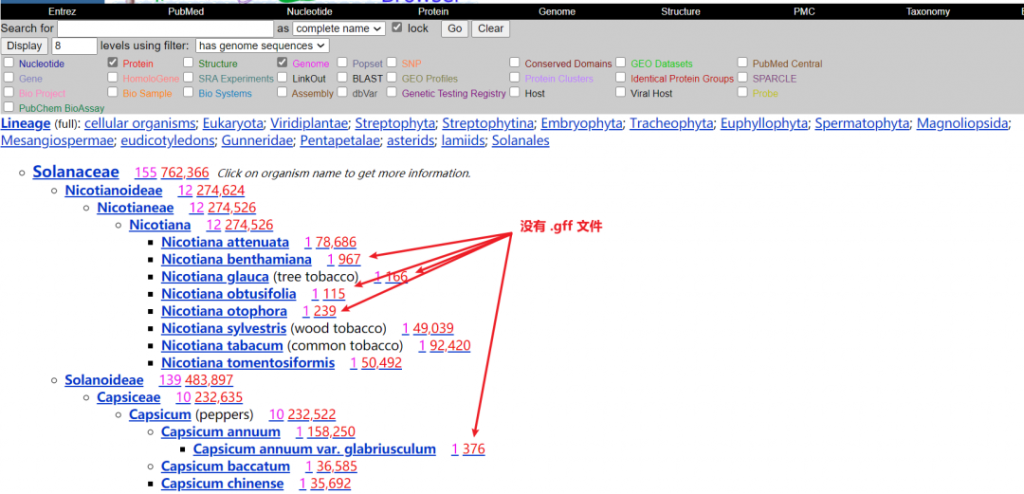
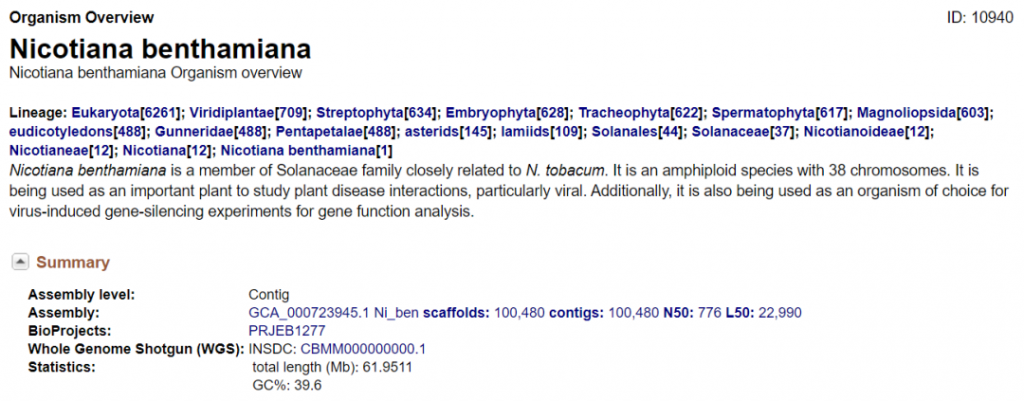
遇到这种情况该怎么办呢?当然是利用科研人的基本功,去PubMed里面查询一下最新的文献,看看是否有人已经完成了该物种的基因组注释。比如实验室最常用到的本氏烟草Nicotiana benthamiana, 从上面的链接点进去的界面是这样的(如下图),根本不存在GFF文件。
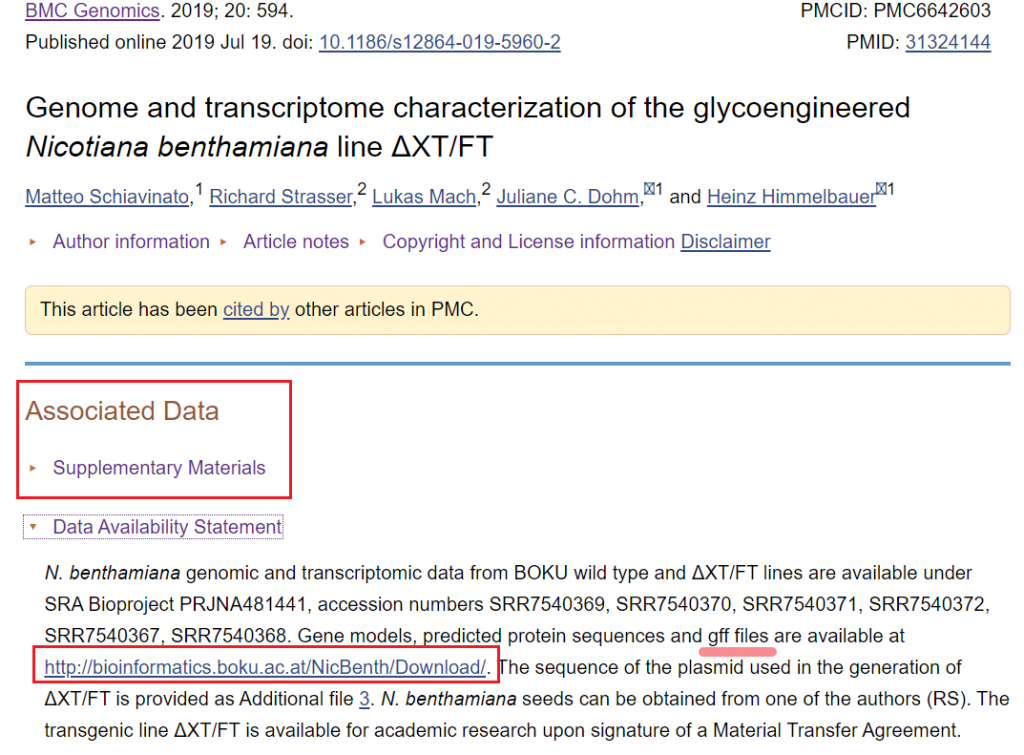
然后,我们在PubMed中以“Nicotiana benthamiana assembly”为关键词进行查询,找到2019年发表在BMC Genomics上的一篇文献,对一个本氏烟草的品种进行了基因组和转录组的特征注释,并提供了GFF文件的链接地址(如下图)。
当然,不是所有的时候都这么幸运,有时文章里明明写了有做基因组注释,但是找遍正文和补充材料都找不到GFF文件的链接地址,这种情况有可能是文章作者考虑到后续研究的需要,暂时没有把数据全部公开。遇到这种情况,不妨试着给文章作者写一封情真意切的邮件,表达一下期望作者能够分享数据信息的请求,有时也会有意外的收获。
以上就是我们为小伙伴们梳理的比较基因组物种基因组信息和基因组注释信息的查询方法,希望能助力大家在基因组研究和比较基因组学的黄金时代实现自己的高光时刻!





