- A+
为了充分利用组学技术的进步,更全面地了解人类疾病,需要新的计算方法来整合分析多种类型的组学数据。美国印第安纳大学的老师2021年6月在《Nature Communications》发表了文章“ MOGONET integrates multi-omics data using graph convolutional networks allowing patient classification and biomarker identification”。是第一个利用图卷积神经网络(GCNs)进行组学数据分类的多组学集成方法。
现状:多组学侧重于无监督的多组学数据整合随着个体化医学的快速发展,带有详细注释的可描述样本表型或性状的数据集越来越广泛。因此,人们对有监督的多组学整合方法越来越感兴趣,这些方法可以识别疾病相关的生物标志物,并对新样本进行预测。早期用于生物医学分类任务的监督数据集成方法包括基于特征串联的策略和基于集成的策略。一方面,基于连接的方法通过直接连接输入数据特征来集成不同的组学数据类型,学习分类模型。另一方面,基于集成的方法集成了来自不同分类器的预测,每个分类器分别在一种组学数据上训练。然而,这些方法未能考虑不同组学数据类型之间的相关性,可能会偏向于某些组学数据类型。
MOGONET是什么?MOGONET是用于生物医学分类任务的有监督的多组学集成框架,也可以称作是多组学数据分类任务的框架。MOGONET的工作流程可以概括为三个部分:(1)预处理。分别对每个组学数据类型进行预处理和特征预选,去除噪声和冗余特征。(2)通过GCNs进行组学特异性学习。针对每种组学数据类型,根据组学特征构建加权样本相似度网络。训练GCN进行组学特异性学习。(3) VCDN多组学集成。使用来自所有组学特定网络的初始类概率预测计算交叉组学发现张量。然后用交叉组学发现张量训练VCDN产生最终预测。mongonet是一个端到端模型,其中组学特定的GCNs和VCDN交替训练直到收敛。

证明MOGONET的能力和多功能性数据集为了验证MOGONET的有效性,我们应用了4个不同的生物医学分类任务,使用4个不同的数据集:阿尔茨海默病(AD)患者和正常对照(NC)的ROSMAP分类、低级别胶质瘤(LGG)的级别分类、肾癌的KIPAN类型分类和乳腺癌浸润性癌(BRCA)的PAM50亚型分类。采用3类组学数据[mRNA表达数据(mRNA)、DNA甲基化数据(meth)和miRNA表达数据(miRNA)]进行分类,提供全面补充的疾病信息。

多组学分类性能评价
为了比较不同的多组学整合方法的有效性,我们随机选取一个数据集中30%的样本作为测试集,剩余70%的样本作为训练集。通过保留原始数据集中的类分布来构建测试集。二分类任务采用正确率(ACC)、F1评分(F1)和受试者工作特征曲线下面积(AUC),多分类任务采用正确率(ACC)、F1评分的支持加权平均值(F1_weighted)和F1评分的宏平均值(F1_macro)。我们在5次不同的随机生成的训练和测试中评估了所有的方法,并报告了这5次实验的评估指标的平均值和标准差。在各种分类任务中,MOGONET优于现有的监督多组学集成方法,并且在各种分类任务中都表现出色

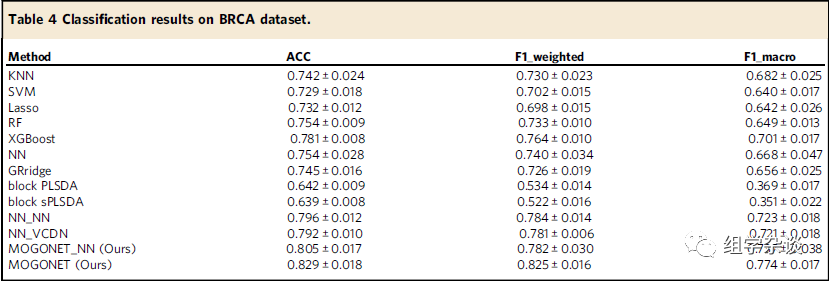
比较了MOGONET与9种现有的组学数据分类算法的分类性能,观察到MOGONET在大多数分类任务中的表现优于其他方法,唯一的例外是在LGG等级分类中,XGBoost和MOGONET的平均AUC相同。MOGONET在不同组学数据类型下的表现:

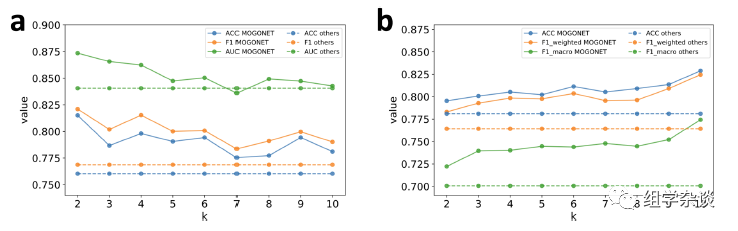
不同超参数k下MOGONET的性能