- A+
所属分类:学术文献
用途
采用GHECOM等算法识别蛋白质/核酸中潜在的小分子结合位点,输出文件可用于分子对接定义口袋。
入口
平台地址:https://cloud.yinfotek.com/
功能入口:左侧菜单栏【计算方案】->【小工具】->【分子结构】->【识别结合位点】
步骤
- 上传受体文件,点击【检测位点】上传处理好的蛋白质/核酸受体结构文件,可参考【处理PDB结构】。
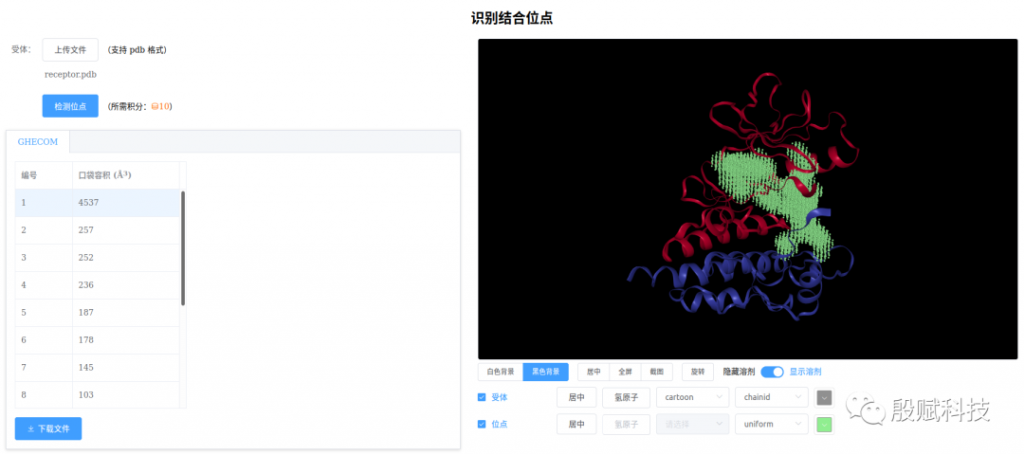
- 浏览位点分布情况表格上方的标签为
算法名称,表中给出了各位点编号和对应的容积。点击某一行,右侧视图即实时显示位点(用一堆小球展示)的位置。1. 通常来说,容积较大的位点更有可能真正的位点。将受体的表现形式改为surface,可更直观展示位点所在的空腔形状。2. 后期会继续增加位点识别算法,通过比较多个算法的位点,提高预测准确度。 - 选择位点,点击【下载文件】该文件可直接用于分子对接定义口袋。

我的微信公共号
我的微信公招扫一扫



