- A+
01 导读
本文针对蓝藻适应其栖息地(淡水,海洋,陆地,寄生环境等)的进化和适应机制,通过比较基因组(650个蓝藻基因组)分析,发现大量的基因表现出生境关联性。陆地蓝藻扩展了其基因家族以应对波动环境,而海洋株蓝藻的基因组相对收缩,以适应贫营养条件;这些基因涉及蓝藻的光合过程、趋化性、营养盐运送或渗透压力,是蓝藻适应特定环境的关键基因,且很大一部分来源于水平基因转移,不同环境下的蓝藻通过获得和保留这些外来基因,获得竞争优势。
02 研究方法
本研究测序了163株蓝藻的基因组(paired-end,100bp,Illumina HiSeq 2000),数据质控后,SPAdes进行组装(kmer长度55,77,100)。此外,作者还从NCBI下载了727个蓝藻基因组,同测序的基因组合并,根据序列质量去除冗余:首先,计算两两基因组间的平均核苷酸相似度(ANI)和alignment fraction值,去除两个值大于99.9%和95%的重复基因组,最终得到650个基因组的数据集,并收集了每一株的元数据(metadata)。此外,作者还附上了包括519株产氧光细菌(Oxyphotobacteria)和7株黑色素杆菌(Melainabacteria),标注为高质量数据集(Cyano HQ)。此后,对基因组数据集进行了注释(KEGG),基于单拷贝基因的种系基因组学(phylogenomic)和比较基因组学分析,以及对基因组大小,光适应基因、基因的生境偏好和水平基因转移(HGT)鉴别的分析。
03 结果与讨论
1)基因组数据提供高解析度的系统发育关系
作者分别基于31个通用核心基因和834个蓝藻普遍通用的单拷贝直系同源基因(Benchmarking Universal Single-copy orthologs,BUSCO)建立系统发育树,发现基因组数据能够展示更高分辨率的系统发育关系(更多的树节点)。
2)生境适应塑造了蓝藻基因组
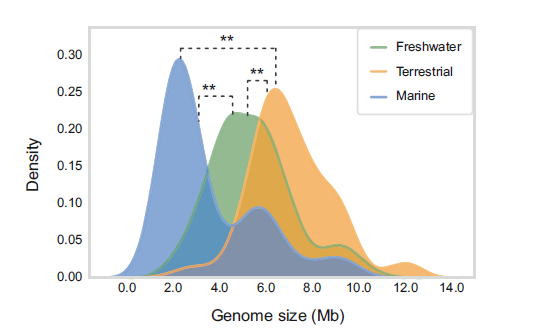
作者将生境特征映射到基因组系统发育树,发现相同的生境常覆盖同一枝的株系。平均来说,陆地蓝藻的基因组要大于海洋和淡水蓝藻,这同陆地生境环境波动更大,需要更为复杂的基因组的假设一致(图1)。富集分析发现,在22个功能基因群中,陆地蓝藻的大多数基因都被富集。
3)生境对光感应、光吸收等基因产生影响
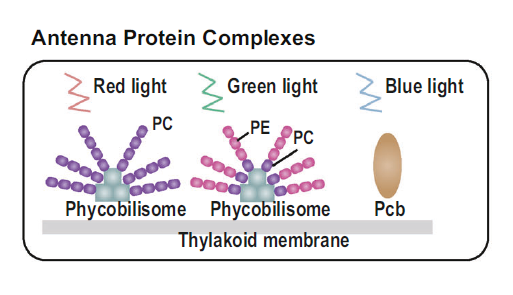
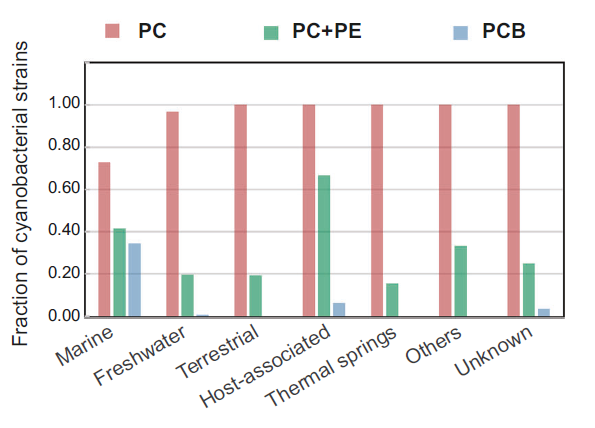
pix基因簇是蓝藻感光蛋白的编码序列,本研究发现,pix基因簇存在于大多数的陆地蓝藻基因组,在大多数淡水和海洋蓝藻中却缺失;光适应的另一策略是改变色素组成。对不同生境蓝藻基因组中的色素合成基因分析发现,海洋蓝藻PC和PE色素的合成能力总体上强于陆地和淡水蓝藻,并且有合成辅助色素的能力,表明其对海洋光照条件的适应(图2)。此外,包括渗透压条件,歧化酶金属组成,营养获取基因在内的很多基因簇都表现出生境差异。例如:抗干旱相关基因(opuACBD)在陆地蓝藻基因组中得到了富集,这同陆地生境的干燥条件相符合。
4)水平基因转移增强了蓝藻的生境适应力
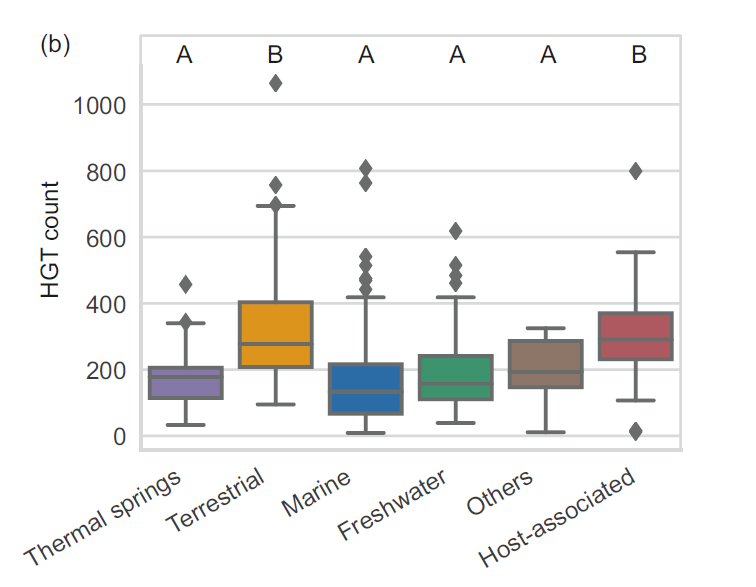
基于BLAST和COUNT软件的方法,在519个高质量基因组中,共检测到近10万个水平基因转移事件,且不同蓝藻基因组间差异明显。宿主分离得到的蓝藻基因组中,HGT事件频率最高,这与宿主的适应过程有关。其次是陆地蓝藻中的HGT,同基因组大小正相关,频率高于海洋和淡水蓝藻(图3)。对这些可能的HGT基因进行注释发现,34%的基因功能未知,51.2%的基因和代谢相关,属于外周和操作基因,而涉及环境信息处理,细胞过程和遗传信息处理功能的核心基因各占约10%。这符合“复杂度假设”,即核心基因相对于外周和操作基因,其转移的概率更低。对不同环境中的HGT基因功能比较发现,很多HGT的存在同生境适应有关:如陆生蓝藻中的xyl B基因,能够帮助蓝藻利用陆地环境中的木质素。
04 总结
本文通过大规模的基因组分析,尝试寻找基因组中适应不同生境的进化痕迹。蓝藻本身作为初级生产者,分布非常广,长期参与地球生物化学过程,这对回答进化适应性问题来说是极好的研究材料。另一方面,本文的研究对象又相对局限,如测序的163株蓝藻,均是来自同一藻种库的淡水和陆地蓝藻,可能存在同化特征。虽然分析结果兼顾了全局性和局部特征(功能基因),但生境划分相对粗糙,更多的是尝试从大生态概念上的解读,文中比较基因组学的方法和工具使用值得借鉴。





